Complexes protéiques, chromatine et maladie
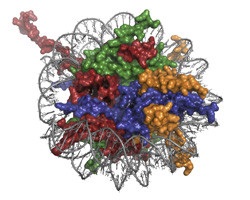
La chromatine, un complexe macromoléculaire d'ADN et de protéines, participe à la régulation des différents processus du métabolisme de l'ADN que sont la réplication, la transcription ou la réparation. Ces processus sont contrôlés par des complexes protéiques qui interagissent avec les nucléosomes, les sous-unités structurelles de la chromatine. Le projet MSL1-MSL3-MOF a été initié pour étudier l'un de ces complexes régulateurs. La protéine MOF est une acétyltransférase qui participe à la transcription de l'ADN. Les deux autres protéines du complexe (MSL1 et MSL3), accroissent l'activité enzymatique de MOF. Le détail des interactions entre d'une part, les composants de ce complexe et d'autre part, la chromatine, demeure inconnu; c'est cette lacune que l'étude a cherché à combler. Les chercheurs ont reconstitué, purifié et caractérisé biochimiquement le complexe protéique afin de cartographier les interactions existantes entre les différents composants du complexe et déterminer son activité catalytique. L'équipe a effectué une première reconstruction tridimensionnelle du complexe de régulation, puis conçu et testé une procédure pour l'assemblage et la purification du substrat de la chromatine. Avant de réaliser une cristallographie aux rayons X du complexe MSL1-MSL3-MOF, les chercheurs ont dû en définir les conditions de cristallisation et d'analyse structurelle en utilisant un système modèle. La cristallographie des grands complexes ADN-protéine est en effet très difficile, les chercheurs ont donc utilisé un complexe protéique bien connu associé au nucléosome, le complexe BAH/nucléosome (bromo-adjacent homology domain) pour en définir les conditions idéales. La structure de BAH qui a été ainsi résolue a révélé l'existence d'une modification conformationelle induite par l'acétylation de l'extrémité N-terminale, une modification post-translationnelle relativement fréquente chez les eucaryotes. Cette nouvelle conformation déplace le domaine BAH et le rapproche du nucléosome comparativement au complexe non acétylé. Cette observation suggère un rôle plus général de la N-acétylation dans les interactions protéiques. Ainsi, l'utilisation de ce système modèle a non seulement permis aux chercheurs de définir les conditions optimales de cristallographie du complexe MOF mais également révélé de nouvelles informations structurelles sur le domaine BAH lui-même. Le dérèglement de nombreux complexes protéiques régulateurs de chromatine étant associés à un état pathologique, la résolution de leurs structures pourrait faciliter la conception de nouvelles molécules thérapeutiques très spécifiques. Les travaux réalisés dans le cadre de ce projet constituent une première étape dans cette direction.