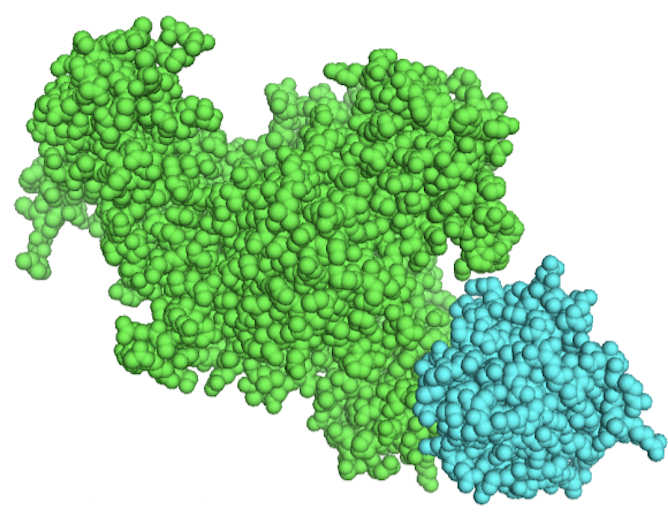
Eine Beleuchtung der Beziehung zwischen der Proteinsequenz, -struktur und -funktion
Nach Wasser sind Proteine die häufigsten Moleküle in unserem Körper. Aus ihnen bestehen Muskeln und anderes Körpergewebe wie unsere Haare. Sie sind ein lebenswichtiger Bestandteil aller lebenden Organismen und dank der Ergebnisse der neuen computergestützten Analyse unter der Führung der Marie-Curie-Stipendiatin Dr. Lucy Colwell werden Wissenschaftler sie besser verstehen und Medikamente entwickeln können. Ihr aus Mathematikern, Chemikern und Biochemikern bestehendes Team an der Universität Cambridge hat Beziehungen zwischen der Sequenz von Proteinen und deren 3D-Struktur sowie Funktionen entdeckt. „Diese Erkenntnisse werden im Mittelpunkt zukünftiger Forschungsbemühungen stehen. Die Beziehung zwischen der Sequenz, Struktur und Funktion von Proteinen ist eine der großen Fragen unserer Zeit“, so Dr. Colwell, die am EVO-COUPLINGS-Projekt beteiligt war. Dank der Ergebnisse von EVO-COUPLINGS könnten Durchbrüche in der Medikamentenentwicklung einfacher erzielt werden, da die Arbeit des Teams Wissenschaftlern dabei hilft, die Tertiärstruktur und Interaktionspartner(öffnet in neuem Fenster) eines Proteins besser vorherzusagen. Die Forschungsarbeit, die mit Unterstützung durch das Marie-Curie-Programm durchgeführt wurde, verwendete einen neuartigen Ansatz, der von den Fachkollegen zunächst skeptisch aufgenommen wurde. Diese Wahrnehmung hat sich jedoch seither geändert. „Ich freue mich darüber, wie massentauglich diese Ideen geworden sind“, sagt sie. „Vor unserer Arbeit war es sehr viel schwieriger, die Proteinstruktur und -interarktionen nur auf Grundlage der Sequenzen vorherzusagen. Dieser Ansatz wird immer mehr zum Standard in diesem Fachbereich und war ein Schlüsselelement zahlreicher aktueller wichtiger Fortschritte.“ Die Lösung der Matrix Unter den Forschern waren auch Mathematiker, die Methoden auf Basis von Zufallsmatrizen entwickelten – ein probabilistischer Ansatz, der von Physikern erarbeitet wurde –, um den Chemikern und Biochemikern die Analyse der Proteinsequenzdaten zu erleichtern. Die experimentellen Verfahren der letzten Jahre haben es Naturwissenschaftlern ermöglicht, große Datenmengen für die Forschung zu sammeln, aber in diesen Informationen etwas Nützliches zu finden, kann einem Kopfschmerzen bescheren. „Meine Arbeit ist, das Wesentliche herauszuarbeiten“, so Dr. Colwell. „Um Proteinsequenzen zur Vorhersage der Struktur zu verwenden, mussten wir zunächst das ‚Rauschen‘ in den Daten identifizieren und herausfiltern, das dadurch verursacht wird, dass verschiedene Proteine miteinander verwandt sind. Dieses Signal musste aus den Daten ‚herausnormalisiert‘ werden, bevor wir mathematische Modelle entwerfen konnten, um nützliche Vorhersagen zu treffen.“ Die Analytiker verwenden Datenvisualisierungsmethoden, um dazu beizutragen, die Struktur in verschiedensten Bereichen zu identifizieren: Diese Methoden können auf jegliche Datensorten angewandt werden. Dr. Colwell arbeitet nun mit Google zusammen, um die Fortschritte im Bereich des maschinellen Lernens dazu zu nutzen, Muster zu erkennen, die für Menschen schwer auszumachen sind.