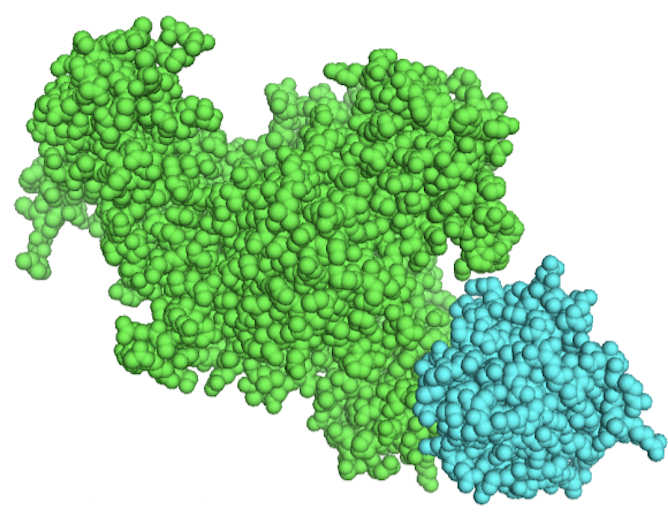
Élucider les relations entre séquence, structure et fonction dans les protéines
Présentes dans les muscles et d’autres tissus corporels tels que les poils, les protéines sont les molécules les plus abondantes dans notre organisme après l’eau. Elles constituent une composante essentielle de tous les organismes vivants et les scientifiques pourront mieux les comprendre et développer des médicaments, grâce à la nouvelle analyse informatique conduite par la Dre Lucy Colwell, titulaire d’une bourse Marie Curie. Son équipe de mathématiciens, de chimistes et de biochimistes de l’Université de Cambridge a découvert des relations entre les séquences des protéines, leurs structures 3D et leurs fonctions. «Ces résultats orienteront les recherches futures. Comprendre les relations entre la séquence, la structure et la fonction des protéines est l’un des grands problèmes de notre époque», explique la Dre Colwell, qui a collaboré au projet Evo-Couplings. EVO-COUPLINGS pourrait permettre de créer plus facilement de nouveaux médicaments, car il améliore la capacité des scientifiques à prédire la structure tertiaire et les partenaires d’interaction(s’ouvre dans une nouvelle fenêtre) d’une protéine. L’étude, entreprise avec le soutien du programme Marie Curie, a adopté une approche novatrice initialement accueillie avec scepticisme par la communauté scientifique. Mais les choses ont changé depuis. «Je suis enthousiasmée de voir à quel point ces idées sont devenues populaires», a-t-elle déclaré. «Avant nos travaux, il était beaucoup plus difficile de prédire la structure et les interactions des protéines à partir des seules séquences. Cette approche est en train de devenir la norme dans le secteur et constitue un élément clé d’un certain nombre de progrès importants récemment réalisés.» Trouver la faille dans la matrice L’équipe de chercheurs comprenait des mathématiciens qui ont élaboré des méthodes faisant appel à la théorie des matrices aléatoires – une approche probabiliste développée par des physiciens – pour aider les chimistes et les biochimistes à analyser les données sur les séquences de protéines. Les récentes techniques expérimentales ont permis aux chercheurs en sciences naturelles de rassembler de grandes quantités de données pour la recherche, mais y trouver celles qui sont utiles relève du casse-tête. «Mon travail consiste à éliminer le superflu», a déclaré la Dre Colwell. «Pour utiliser les séquences protéiques afin de prédire la structure, nous avons d’abord dû identifier et éliminer le “bruit” dans les données dû au fait que des protéines différentes sont liées les unes aux autres. Ce signal doit être “normalisé” à partir des données avant de pouvoir construire des modèles mathématiques permettant de faire des prédictions utiles.» Les analystes utilisent des méthodes de visualisation des données pour faciliter l’identification de la structure dans une gamme de champs: ces méthodes peuvent être appliquées à tous les types de données. La Dre Colwell travaille maintenant avec Google pour utiliser les avancées réalisées dans l’apprentissage automatique afin de distinguer les modèles difficiles à identifier par les humains.