Strumenti migliori per spiegare la dinamica delle proteine di membrana
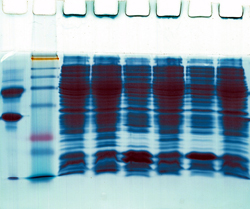
La dinamica delle proteine è collegata alla funzione, all'aggregazione e al ripiegamento delle proteine e la spettroscopia a risoluzione temporale è particolarmente adatta a rivelare queste dinamiche. La dinamica delle proteine si manifesta anche quando viene applicata una perturbazione rapida. Un esempio è quando un impulso laser viene applicato alle proteine con fotocicli. Sia negli esperimenti con scambio idrogeno/deuterio (HDX) che in quelli con perturbazione, la risposta a risoluzione temporale del sistema è un processo di rilassamento multiesponenziale. Le informazioni da caratterizzare sono il numero, il valore e la natura delle costanti di velocità del processo di rilassamento, non il processo stesso. Il progetto NAGOYA2BCN puntava a migliorare, sviluppare e applicare metodi di massima entropia e bayesiani per analizzare i dati multiesponenziali ottenuti nello HDX delle proteine di membrana e nei fotocicli delle proteine di membrana. Al contempo ha iniziato a usare nuovi approcci HDX sperimentali per ottenere le informazioni sulla dinamica delle proteine di membrana mediante spettroscopia a infrarossi (IR) e applicandoli ad almeno tre sistemi di proteine di membrana: il transporter di melibiosio, la batteriorodopsina e una chimera di recettori accoppiati alle proteine G. I risultati comunicati indicano che la scala temporale delle fluttuazioni proteiche che si possono monitorare e caratterizzare aumenta in riferimento a una finestra temporale maggiore coperta negli esperimenti HDX. Nonostante siano stati incontrati alcuni ostacoli nella progettazione di un apparato sperimentale per la spettroscopia IR per migliorare la finestra temporale degli esperimenti HDX, l'equipe di ricerca ha sviluppato un approccio semplice che cambia la proteina da un H2O medio a un 95% D2O medio in pochi secondi. Sono stati fatti progressi anche durante la collaborazione con il Prof. Kandori a Nagoya, Giappone, sull'interferenza bayesiana con campionamento Markov Chain Monte Carlo. In altre parole sono stati fatti passi avanti per migliorare/sviluppare i diversi strumenti e software che facilitano l'analisi dei dati HDX e del fotociclo bR. Questi miglioramenti sono stati inclusi in un programma visivo eseguito in Matlab. I successi del progetto NAGOYA2BCN offrono informazioni uniche e fondamentali per comprendere meglio la dinamica delle proteine di membrana.