Osservare l'aprirsi del DNA in tempo reale
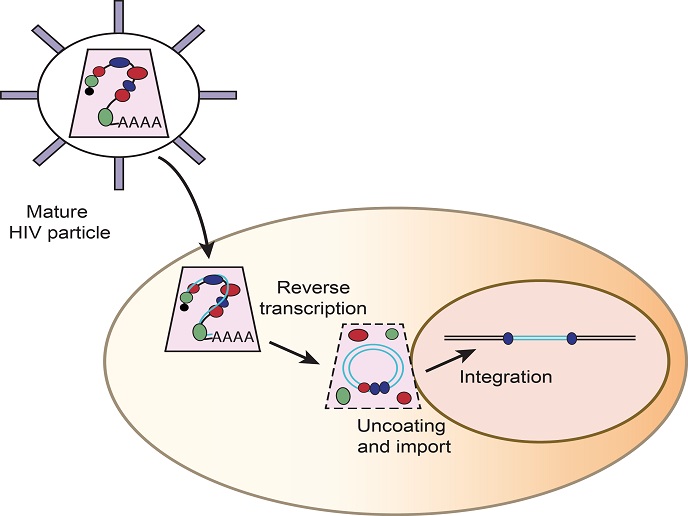
L'acido deossiribonucleico a doppio filamento (dsDNA) che forma il genoma si compone di sequenze corrispondenti a geni specifici nonché di sequenze nonsenso. Ora gli scienziati sanno dove iniziano e finiscono tutti i geni, ma sono ancora lontani dalla fine dell'attività parimenti scoraggiante di caratterizzare integralmente come siano prodotte le proteine, come sia possibile modulare i passaggi e, ovviamente, quale ruolo svolgano le proteine negli stati di malattia e salute. Le cellule si affidano a un processo bifase di trascrizione e traduzione al fine di "leggere" la sequenza di DNA di un gene e quindi tradurlo nella produzione di una proteina. La trascrizione implica la separazione del dsDNA nel nucleo. Il suo avvio richiede la polimerasi dell'acido ribonucleico (RNA), una specie di molecola "un-zipper" (che apre a cerniera). I ricercatori finanziati dall'UE che hanno lavorato nell'ambizioso progetto SM-Transcription ("A single-molecule view of initial transcription") hanno cercato di combinare la diagnostica per immagini spettroscopiche di singola molecola con un righello spettroscopico su nanoscala, per visualizzare modifiche estremamente piccole di complessi di DNS che segnalano l'avvio della trascrizione. Il sistema di modello prescelto è stato costituito dai batteri dell'Escherichia coli (E. coli). Anche se molti processi biologici si affidano sull'unzipping (apertura) e/o sullo zipping (chiusura) di piccoli frammenti di dsDNA, attualmente sono disponibili pochissime tecniche capaci di analizzare tale procedura in tempo reale a livello di singola molecola. Il team di SM-Transcription ha sviluppato un nuovo test modificato di trasferimento di energia per risonanza Förster (Förster resonance energy transfer - FRET) per monitorare un frammento di DNA e indicare se fosse a filamento doppio o unico. Il FRET modificato (smorzato) (quFRET – quenched FRET) si basa su due molecole fluorescenti, una su ciascuno dei due filamenti del DNA che presentano un quenching (diminuzione) del segnale fluorescente al contatto. Servendosi del test quFRET, gli scienziati hanno rilevato l'avvio fallito da parte della polimerasi del RNA e hanno chiarito la modalità d'azione di due potenti inibitori dell'inizio della trascrizione. Inoltre hanno chiarito una serie di meccanismi e cambiamenti conformazionali correlati alla polimerasi del RNA nell'E. coli e hanno fornito le prove sperimentali delle fluttuazioni conformazionali attorno al millisecondo utilizzando righelli FRET. Le implicazioni del nuovo test sviluppato dal team SM-Transcription sono impressionanti. Un completo ripasso dell'intero genoma umano e del processo di trascrizione di ciascuna proteina rappresenta solo l'inizio.