Determinando la dinámica ribosomal
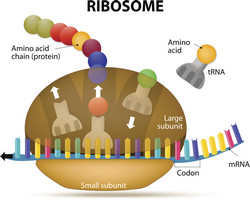
La síntesis de proteínas en las células tiene lugar en unas estructuras macromoleculares especializadas denominadas ribosomas, que están constituidas por ARN y proteínas. Aunque análisis de alta resolución han proporcionado una imagen precisa de la estructura del ribosoma, aún no se conoce en detalle la dinámica de las interacciones proteína-ARN. Esto se debe fundamentalmente a la inestabilidad de estos complejos in vitro. Los investigadores del proyecto financiado por la Unión Europea RSDYN (Ribosome dynamics analysed by novel cross-linking/mass spectrometry) se propusieron abordar este reto por medio de nuevas técnicas basadas en la espectrometría de masas con el fin de determinar la interacción entre proteínas y ARN. En sus experimentos, los socios de RSDYN irradiaron células vivas de levadura e identificaron la parte del péptido señal que se unía con el ARN. Gracias a este método, estos lograron localizar de manera precisa los sitios de unión a ARN en cientos de proteínas de levadura. Esta información fue combinada con análisis de alta resolución de estructuras ribosomales de levadura y de complejos individuales de proteína-ARN. Los investigadores también llevaron a cabo análisis de genoma completo de dianas de ARN para la enolasa, una nueva proteína de unión a ARN. Para determinar interacciones proteína-proteína en complejos ribosomales, los socios del consorcio emplearon cepas de Escherichia coli defectuosas para el ensamblaje de ribosomas y estudiaron la alteración de la composición y de la estructura del ribosoma. El método desarrollado por el proyecto RSDYN complementa las estrategias existentes basadas en la cristalografía de rayos X y en la resonancia magnética nuclear (NMR) empleadas para el estudio de la dinámica de ribonucleopartículas como los ribosomas. Este método debería ser de gran utilidad para una gran variedad de campos de investigación, desde la biología molecular básica y el diseño de nuevos fármacos hasta el estudio de las interacciones proteína-ADN. Es más, los resultados de este estudio ayudarán al desarrollo de la próxima generación de fármacos antibacterianos, que actúan de manera específica frente a factores de unión en la biogénesis ribosomal.