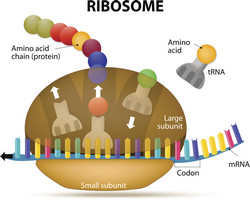
Chiarire le dinamiche dei ribosomi
La sintesi delle proteine nelle cellule avviene in strutture macromolecolari specializzate dette ribosomi, composti da RNA e proteine. Sebbene l’analisi ad alta risoluzione abbia fornito un quadro dettagliato della struttura dei ribosomi, la dinamica dell’interazione proteine-RNA resta per lo più sconosciuta, principalmente a causa dell’instabilità dei complessi in vitro. Gli scienziati del progetto RSDYN (Ribosome dynamics analysed by novel cross-linking/mass spectrometry), finanziato dall’UE, hanno deciso di affrontare questa sfida tramite nuove tecnologie basate sulla spettrometria di massa e di determinare il cross linking tra proteine e RNA. Nei loro esperimenti i membri del consorzio RSDYN hanno irradiato cellule vive di lievito e identificato la parte del peptide che interagiva con l’RNA. Con questo approccio hanno mappato in modo preciso i siti di legame all’RNA di centinaia di proteine del lievito. Queste informazioni sono state combinate con analisi ad alta risoluzione delle strutture dei ribosomi del lievito e dei singoli complessi proteina-RNA. I ricercatori hanno svolto anche analisi genome-wide dei target di RNA della nuova proteina enolasi che lega l’RNA. Per chiarire le interazioni proteina-proteina nei complessi ribosomiali il consorzio ha utilizzato ceppi di Escherichia coli difettivi nell’assemblaggio dei ribosomi, e ha studiato le alterazioni nella composizione e nella struttura dei ribosomi. Il metodo RSDYN completa le strategie esistenti basate su cristallografia a raggi X e RMN per lo studio delle dinamiche di ribonucleoparticelle come i ribosomi. Questo metodo dovrebbe essere molto utile in numerose aree di ricerca, dalla biologia molecolare di base alla progettazione dei farmaci allo studio delle interazioni tra proteine e DNA. I risultati di questo studio favoriranno inoltre lo sviluppo di farmaci antibatterici di prossima generazione che bersagliano i fattori di assemblaggio nella biogenesi dei ribosomi.