Innovative Techniken für die molekulare Simulation
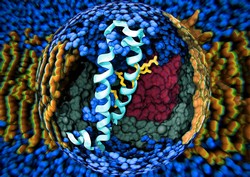
Das EU-finanzierte Projekt ERIKLINDAHLERC2007 (Multiscale and distributed computing algorithms for biomolecular simulation and efficient free energy calculations) hat den Stand der Technik im Bereich der biomolekularen Simulation vorangetrieben. Die Forscher erhöhten erfolgreich die Leistungsfähigkeit solcher Werkzeuge um ein Vielfaches und verbesserten deren Vorhersagegenauigkeit. Die Forscher machten bemerkenswerte Fortschritte, angefangen bei der Entwicklung der Version 4.6 von GROMACS(öffnet in neuem Fenster). Diese Software kann verschiedene Arten von Biomolekülen (z.B. Proteine, Lipide und Nukleinsäuren) zusammen mit ihren entsprechenden Kraftfeldern über CHARMM27-spezifische Funktionen verarbeiten. Sie zeigten eine etwa 40-fach höhere Leistung und eine zehnfache Verbesserung der Parallelisierung für Simulationen. Außerdem war dieser frei verfügbare Simulationscode fünfmal schneller als der aktuelle Stand der Technik. Die Umsetzung von freien Energieberechnungen auf der Grundlage der "Bennett Acceptance Ratio" ermöglichte die on-the-fly-Analyse mit verbesserter Genauigkeit auch in verteilten und in Cloud-Rechenumgebungen. Darüber hinaus automatisierten die Projektmitglieder erfolgreich die Schaffung von Eingangsdaten der freien Energie während statistische Auswertungen Standardfehlerabschätzungen für alle Berechnungen der freien Energie lieferten. Das Tüpfelchen auf dem i war jedoch die Entwicklung eines neuen Rahmens für "parallele adaptive Molekulardynamik", der verteiltes Rechnen mit Molekulardynamik-Simulationen kombiniert. Diese auf Copernicus(öffnet in neuem Fenster) verfügbare Open-Source-Tool terminiert automatisch Tausende von eng gekoppelten Simulationen, während eine verteilte Rechenumgebung die Sampling-Effizienz um das Zigfache verbessert. Adaptive Simulationen für freie Energieberechnungen und die automatisierte Erstellung von Simulationsparametern für kleine Moleküle haben dazu beigetragen, die Anzahl der erforderlichen Simulationen zu reduzieren. Als Ergebnis kann Copernicus Solvatation und die Bindung freier Energien einer großen Reihe von Verbindungen innerhalb weniger Stunden bestimmen. Darüber hinaus kostet dies weniger als ein Euro pro Verbindung. Diese neuen Werkzeuge halfen den Forschern, Simulationen von wichtigen biologischen Systemen durchzuführen, etwa von gesteuerten Ionenkanälen und Membranproteinen. Zentrale Erkenntnisse führten zu mehreren Veröffentlichungen in Zeitschriften mit erheblichen Auswirkungen für die Entwicklung von Arzneimitteln. Zum Beispiel lieferte die Untersuchung von Liganden-gesteuerten Ionenkanälen neue Einblicke in den allosterischen Dual-Site-Modulationsmechanismus. Dies könnte sich für die Entwicklung neuartiger Paare von Ein-/Aus-Anästhetika als nützlich erweisen. Insgesamt konnten die Werkzeuge und Erkenntnisse von ERIKLINDAHLERC2007 unser Wissen über degenerative Krankheiten wie Creutzfeldt-Jakob verbessern und das rationale Design von Medikamenten unterstützen. Die Anwendung dieser Methoden könnte auch über die Biomedizin hinaus auf Bereiche der Polymerwissenschaft und der Nanotechnologie ausgeweitet werden.