Nowe zaawansowane techniki symulacji cząsteczkowej
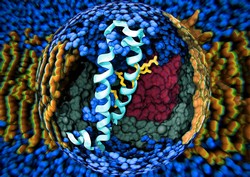
W ramach finansowanego ze środków UE projektu ERIKLINDAHLERC2007 (Multiscale and distributed computing algorithms for biomolecular simulation and efficient free energy calculations) poczyniono znaczące postępy w zakresie symulacji biocząsteczkowych. Badacze z powodzeniem zwiększyli wydajność tych narzędzi o kilka rzędów wielkości i poprawili dokładność przewidywania. Badacze poczynili zdumiewające postępy, rozpoczynając od opracowania oprogramowania GROMACS(odnośnik otworzy się w nowym oknie), wersja 4.6. To oprogramowanie przeznaczone jest dla różnych biocząsteczek (np. białek, lipidów i kwasów nukleinowych) oraz odpowiednich pól sił za pomocą funkcji CHARMM27. Naukowcy zaprezentowali około 40 razy większą wydajność oraz dziesięciokrotną poprawę obliczania równoległego dla symulacji. Ponadto ten dostępny bezpłatnie kodu symulacji był pięciokrotnie szybszy niż aktualnie używany. Zastosowanie obliczeń wolnej energii na podstawie wskaźnika akceptowalności Benetta umożliwiło dokładniejszą analizę w czasie rzeczywistym nawet w rozproszonych środowiskach obliczeniowych i środowiskach opartych o chmurę. Co więcej, uczestnicy projektu zautomatyzowali tworzenie danych wejściowych dotyczących wolnej energii, a analizy statystyczne umożliwiły wyeliminowanie standardowych błędów ze wszystkich obliczeń wolnej energii. Głównym osiągnięciem było jednak opracowanie nowatorskich ram umożliwiających równoległą adaptacyjną dynamikę molekularną łączącą rozproszone systemy obliczeniowe z symulacjami dynamiki molekularnej. To narzędzie typu open source dostępne w witrynie Copernicus(odnośnik otworzy się w nowym oknie) automatycznie planuje tysiące silnie powiązanych symulacji, a rozproszone systemy obliczeniowe zwiększają jakość próbkowania o kilka rzędów wielkości. Symulacje adaptacyjne obliczeń wolnej energii oraz zautomatyzowane tworzenie parametrów symulacji dla małych cząsteczek pozwoliło na obniżenie liczby wymaganych symulacji. W rezultacie program Copernicus umożliwił określenie solwatacji oraz wiązanie wolnej energii dużych serii związków w ciągu kilku godzin. Ponadto koszt tych procesów jest niższy niż euro na związek. Nowe narzędzie umożliwiło badaczom przeprowadzenie symulacji ważnych systemów biologicznych, takich jak bramkowe kanały jonowe oraz białka błonowe. Dokonano kluczowych odkryć o istotnym wpływie na tworzenie leków, które zostały opublikowane w magazynach branżowych. Na przykład, badanie kanałów jonowych bramkowanych ligandem udostępniło nowe informacje na temat mechanizmu allosterycznych modulacji podwójnych. Zdobyta wiedza może być użyteczna w projektowaniu nowych par środków znieczulających. Podsumowując, narzędzia i wnioski z badania ERIKLINDAHLERC2007 mogą pogłębić wiedzę dotycząca chorób zwyrodnieniowych, takich jak choroba Creutzfeldta-Jakoba, oraz pomóc w racjonalnym projektowaniu leków. Oprócz biomedycyny, metody te mogą zostać zastosowane w nauce o polimerach i nanotechnologii.