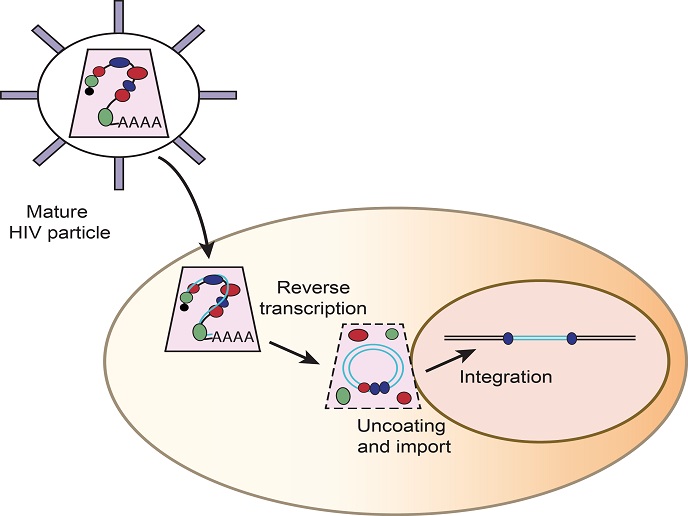
L’evoluzione della resistenza farmacologica
Le cellule hanno una capacità stupefacente di adattarsi alle perturbazioni genetiche e ambientali, che spesso si traduce nella riorganizzazione delle loro reti metaboliche o in modifiche della regolazione e dell’espressione genetica. I dettagli di questi processi, a livello sia molecolare sia evolutivo, sono però ancora poco noti. Sono infatti necessari studi di laboratorio sistematici e su larga scala che approfondiscano l’evoluzione dell’ipersensibilità e della resistenza incrociata batterica in condizioni controllate. Per affrontare questo problema, gli scienziati del progetto NETWORK EVOLUTION (Integrated evolutionary analyses of genetic and drug interaction networks in yeast), finanziato dall’UE, hanno utilizzato il batterio Escherichia coli, applicando una metodologia integrata basata sulla biologia dei sistemi volta a decifrare la resistenza incrociata tra gli antibiotici. Hanno quindi combinato approcci di laboratorio ad alto rendimento, il sequenziamento dell’intero genoma e la biologia computazionale per interpretare gli esperimenti evolutivi e studiare i ceppi resistenti sviluppati in laboratorio. I ricercatori hanno presentato la prima mappa sperimentale della resistenza incrociata e delle interazioni di sensibilità collaterale, i cui risultati rivelano che due antibiotici sono evolutivamente collegati se l’adattamento a uno dei due cambia la sensibilità all’altro. Questa rete era coerente con i dati di co-resistenza su larga scala di isolati clinici della stessa specie, a conferma della rilevanza medica di questa risorsa unica. L’analisi della mappa evolutiva ha identificato i principi generali che governano l’evoluzione dei modelli di resistenza incrociata, con un risultato che apre la strada a metodi in silico per la previsione della propensità alla resistenza incrociata di nuovi composti antimicrobici. Il consorzio, inoltre, ha identificato un meccanismo molecolare alla base dell’ipersensibilità dei batteri antibiotico-resistenti ad altri agenti, che rappresenterà senza dubbio un aiuto nello studio razionale di terapie multi-farmaco sequenziali. Complessivamente, il progetto NETWORK EVOLUTION ha affrontato problematiche chiave dell’adattamento microbico agli antibiotici, mostrando soprattutto che è possibile prevedere modelli di evoluzione che a lungo termine aiuteranno a prendere decisioni informate in medicina.