Las matemáticas de la evolución del genoma
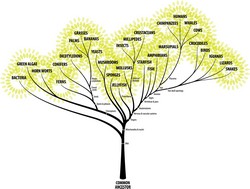
La noción de que todas las formas de vida están relacionadas genéticamente es una de las nociones más románticas de la ciencia. Se llama filogenia de los organismos e implica que las distintas especies proceden de formas anteriores de las que descienden. Además, todos los organismos están relacionados por la transmisión de genes a lo largo de las ramas del árbol de la vida. Las hojas de este inmenso árbol evolutivo corresponderían a los organismos vivos en la actualidad. Las raíces representan los últimos ancestros comunes de todas las especies del árbol. Se cree que la información genética (incluido el ADN y las proteínas) evolucionó desde las raíces hasta las hojas según modelos de Markov generales. Los científicos estudiaron los modelos de Markov generales en el marco del proyecto TREEMODELS (Algebraic statistics of general Markov models). Esta clase de modelos contiene muchas simulaciones que se utilizan en filogenética para explicar los parecidos entre plantas, animales y microorganismos. Estos modelos estadísticos son algebraicos porque se pueden definir en términos de restricciones o parametrizaciones polinómicas. El análisis de los modelos de Markov generales se basó en el uso de geometría algebraica que permite representar las independencias entre distintas variables. Las estadísticas algebraicas ofrecieron un marco de computación para abordar problemas como el intercambio de marcadores que dificultan la interpretación de los resultados. El equipo de TREEMODELS también propuso modelos estadísticos alternativos más sencillos de los procesos que generan datos a partir de los genomas y que, mediante lo que se conoce como inferencia estadística, han permitido sacar conclusiones sobre estos procesos. Las secuencias genómicas son como los planos de la vida, pero su funcionamiento y evolución apenas se conocen todavía. Estos nuevos modelos, llamados supermodelos marginales, permitieron representar la evolución biológica con mayor eficacia que los modelos de árbol filogenético convencionales. El equipo se aseguró de que los parámetros de los árboles fueran identificables para pudieran derivarse las historias evolutivas de manera coherente. Los científicos aportaron nuevos conocimientos sobre la geometría de los distintos modelos de árbol estudiando diferentes variedades algebraicas. Empleando cumulantes de árboles, demostraron que la variedad secante de la variedad Segre es tórica. También se prestó una atención especial a las intensas propiedades de positividad y convexidad de otra variedad algebraica proyectiva llamada variedad exponencial. La estadística algebraica es un campo nuevo cuyo alcance todavía se está expandiendo. El proyecto TREEMODELS avanzó por el camino de la estadística algebraica con la finalidad de desarrollar herramientas de inferencia necesarias para el análisis de secuencias biológicas. Se espera que de su labor surja una infraestructura computacional rigurosa para organizar los conocimientos biológicos.