Les mathématiques de l'évolution du génome
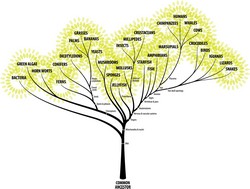
La notion que toutes les formes de vie sont apparentées génétiquement est l'une des plus romantiques de la science. La notion de phylogénie des organismes signifie qu'une espèce vivante descend de formes différentes, qui l'ont précédée. En outre, tous les organismes sont reliés par la transmission des gènes le long des ramifications de l'arbre de la vie. Les feuilles portées par les branches de ce grand arbre de l'évolution sont les organismes qui vivent aujourd'hui. Les racines représentent le plus récent ancêtre commun à toutes les espèces présentes dans l'arbre. Des chercheurs supposent que l'information génétique (l'ADN et les protéines) a évolué depuis les racines en suivant des modèles de Markov généraux. Les scientifiques du projet TREEMODELS (Algebraic statistics of general Markov models) ont étudié les modèles de Markov généraux. Cette catégorie comprend de nombreux modèles utilisés en phylogénétique pour expliquer les similitudes entre les plantes, les animaux et les micro-organismes. Ces modèles sont statistiques et algébriques car ils peuvent être définis par des contraintes polynomiales ou des paramétrisations. L'analyse des modèles de Markov généraux s'est appuyée sur une géométrie algébrique, qui permet de représenter les indépendances entre diverses variables. Les statistiques algébriques apportent un cadre de calcul pour gérer des problèmes comme la commutation d'étiquettes, qui rendent difficiles l'interprétation des résultats. L'équipe de TREEMODELS a aussi proposé d'autres modèles statistiques des processus, plus simples, pour générer des données à partir des génomes et tirer des conclusions de ces processus (par inférence statistique). Les séquences du génome sont le plan d'un organisme et de la vie, mais leurs fonctions et leur évolution restent mal comprises. Ces nouveaux modèles, appelés supermodèles marginaux, semblent représenter l'évolution biologique plus efficacement que les modèles standards d'arbre phylogénétique. L'équipe s'est assurée que les paramètres des arbres étaient identifiables afin que les histoires évolutionnaires soient inférées de façon cohérente. Les scientifiques ont apporté de nouvelles connaissances sur la géométrie des différents modèles d'arbres en étudiant différentes variétés algébriques. À l'aide de cumulants d'arbres, ils ont prouvé que la variété sécante de la variété Segre est torique. L'accent a également été mis sur la forte positivité et les propriétés de convexité d'une autre variété algébrique prévue, appelée variété exponentielle. Les statistiques algébriques sont un nouveau domaine, dont la portée continue de s'élargir. Le projet TREEMODELS s'est engagé dans la voie des statistiques algébriques afin de réaliser les outils nécessaires à l'analyse des séquences biologique. À son terme, il devrait avoir produit un cadre de calcul rigoureux pour organiser les connaissances en biologie.