Matematyczne ujęcie ewolucji genomu
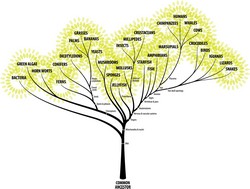
Idea pokrewieństwa genetycznego wszystkich form życia jest jedną z najbardziej romantycznych w nauce. Ta tak zwana filogeneza organizmów głosi, że poszczególne gatunki pochodzą od poprzednich form. Dodatkowo, wszystkie organizmy są powiązane poprzez geny przekazywane wzdłuż gałęzi drzewa filogenetycznego. Liście tego rozległego drzewa ewolucyjnego odpowiadają organizmom, które żyją obecnie. Korzenie reprezentują ostatnich wspólnych przodków wszystkich gatunków w obrębie drzewa. Naukowcy założyli, że informacja genetyczna dotycząca DNA i białek wyewoluowała od korzeni aż po liście według ogólnych modeli Markowa. W ramach projektu TREEMODELS (Algebraic statistics of general Markov models) naukowcy badali ogólne modele Markowa. Ta grupa modeli zawiera wiele symulacji używanych w filogenetyce do wyjaśniania podobieństw między roślinami, zwierzętami i mikroorganizmami. Są to algebraiczne modele statystyczne, więc można je określić pod względami polinominalnych więzów lub parametryzacji. Analiza ogólnych modeli Markowa bazowała na użyciu geometrii algebraicznej, która pozwala na prezentację niezależności różnych zmiennych. Statystyka algebraiczna zapewnia obliczeniowy plan ramowy do rozwiązania takich problemów, jak komutacja etykietami (ang. label switching), utrudniająca interpretację wyników. Zespół projektu TREEMODELS zaproponował również alternatywne, prostsze modele statystyczne procesów generujących dane z genomów i umożliwiających tak zwane wnioskowanie statystycznie o tych procesach. Sekwencje genomów to szablony dla form życia, a ich ewolucja i funkcje pozostają słabo wyjaśnione. Te nowe modele, tzw. supermodele graniczne, lepiej odzwierciedlają ewolucję biologiczną niż standardowe drzewa filogenetyczne. Zespół zadbał o identyfikowalność parametrów drzewa, przez co można wyciągać spójne wnioski dotyczące przebiegu ewolucji. Naukowcy poczynili postępy w określaniu geometrii różnych modeli drzew, badając różne zmienne algebraiczne. Korzystając z kumulant drzew wykazali, że rozmaitość siecznej rozmaitości Segrego jest toryczna. Prace dotyczyły też silnej pozytywności i wypukłości innej rozmaitości algebraicznej, znanej jako rozmaitość wykładnicza. Statystyka algebraiczna jest nową dziedziną, której zakres ciągle się rozszerza. Uczestnicy projektu TREEMODELS korzystali ze statystyki algebraicznej, aby stworzyć narzędzia do wnioskowania na potrzeby analizy sekwencji biologicznych. Wynikiem tego projektu będzie ścisły obliczeniowy plan ramowy do porządkowania wiedzy biologicznej.