La matematica dell’evoluzione del genoma
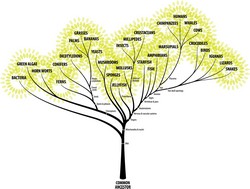
La nozione che tutte le forme di vita siano geneticamente collegate, è una delle nozioni più romantiche della scienza. Questa cosiddetta filogenia degli organismi implica che specie differenti discendano da forme precedenti. Inoltre tutti gli organismi sono collegati dal passaggio dei geni lungo i rami dell’albero della vita. Le foglie di questo ampio albero evolutivo corrispondono agli organismi attualmente viventi. Le radici rappresentano gli ultimi antenati comuni di tutte le specie nell’albero. Gli scienziati ipotizzato che le informazioni genetiche, tra cui il DNA e le proteine, si siano evolute dalle radici alle foglie secondo i modelli generali di Markov. Nell’ambito del progetto TREEMODELS (Algebraic statistics of general Markov models) gli scienziati hanno studiato i modelli generali di Markov, una classe di modelli che contiene molte simulazioni utilizzate in filogenetica per spiegare le somiglianze tra piante, animali e microrganismi. Questi modelli statistici sono algebrici in quanto possono essere definiti in termini di vincoli polinomiali o parametrizzazioni. L’analisi dei modelli generali di Markov era basata sull’impiego della geometria algebrica che permette la rappresentazione di indipendenze tra diverse variabili. La statistica algebrica offriva un quadro computazionale per affrontare problemi quali la commutazione di etichetta (label switching) che rendono difficile l’interpretazione dei risultati. Il team TREEMODELS ha anche proposto modelli statistici più semplici e alternativi dei processi che generano dati dai genomi e, in quella che è nota come inferenza statistica, ha tratto conclusioni su questi processi. Le sequenze genomiche sono la mappa della vita, ma la loro funzione ed evoluzione è poco conosciuta. Questi nuovi modelli, chiamati supermodelli marginali, sembrano rappresentare l’evoluzione biologica in modo più efficace rispetto ai modelli ad albero filogenetico standard. Il team ha garantito che i tre parametri fossero identificabili affinché le storie evoluzionistiche possano essere desunte in modo coerente. Gli scienziati hanno offerto maggiori indizi sulla geometria dei diversi modelli di albero della vita studiando diverse varietà algebriche. Usando cumulanti di alberi, hanno dimostrato che la varietà secante della varietà di Segre è torica. È stata anche prestata attenzione alla forte positività e proprietà di convessità di un’altra varietà algebrica proiettiva, nota come varietà esponenziale. La statistica algebrica è un nuovo settore il cui campo d’azione si sta ancora ampliando. Il progetto TREEMODELS si è mosso lungo il percorso dell’algebra statistica con l’obiettivo di sviluppare strumenti di inferenza necessari per analisi di sequenze biologiche. Ci si attende che emerga un rigoroso quadro computazionale per l’organizzazione del sapere biologico.