Beitrag bakterieller Ökosysteme zur Resistenzbildung bei Mikroorganismen
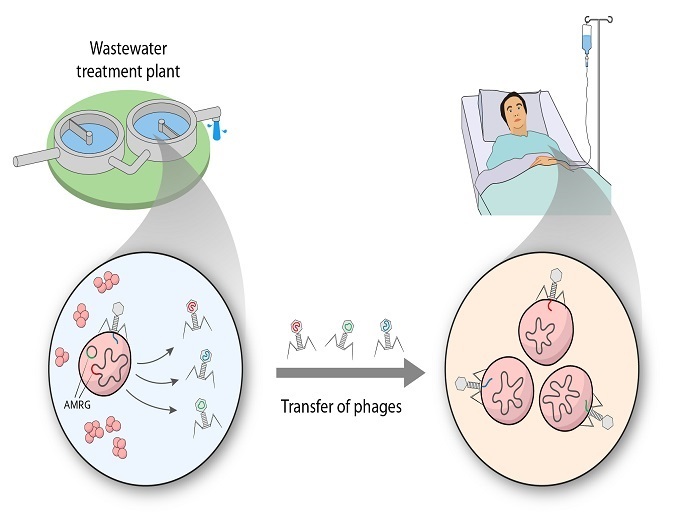
Bakteriophagen spielen als häufigste Organismen auf der Erde für die mikrobielle Physiologie und das Gleichgewicht von Ökosystemen eine zentrale Rolle. Da sie artspezifisch sind und keinen Einfluss auf die menschliche Kommensalflora (u. a. Darmbakterien) haben, sind sie eine vielversprechende Alternative zu Antibiotika im Kampf gegen pathogene Bakterien. Neuere Studien deuten bei Phagen jedoch auf ein breiteres Wirtsspektrum hin als bislang angenommen, und indirekt wird auch davon ausgegangen, dass Bakteriophagen an der Übertragung von AMR-Genen beteiligt sind.
Forschung an Bakteriophagen in vom Menschen geschaffener Umgebung
Finanziert über die Marie-Skłodowska-Curie-Maßnahmen (MSC) untersuchte das Projekt ROPHARE, inwieweit Bakteriophagen am horizontalen Gentransfer von AMR-Genen zwischen Bakterien beteiligt sind. Die Arbeitshypothese lautete, dass nicht allein der missbräuchliche Einsatz von Antibiotika für die weltweite Zunahme antimikrobieller Resistenzen verantwortlich ist, „sondern es besteht ein zunehmender Konsens über eine Kooperation zwischen Mikrobengemeinschaften, Antibiotika und AMR-Genen in verschiedenen Ökosystemen bei der Weitergabe von AMR-Genen und damit der Ausbreitung von Resistenzen“, erklärt die MSC-Stipendiatin Elena Gómez-Sanz. Um diese komplexe Wechselwirkung zu analysieren, wurden zwei Umweltsysteme betrachtet, die vom Menschen geschaffen wurden: landwirtschaftlich genutzte Bodenflächen und eine Kläranlage. Hierfür isolierten und charakterisierten die Forschenden in (unbehandeltem und behandeltem) Klärwasser etwa 100 neuartige Staphylokokken-Bakteriophagen und extrahierten Genmaterial aus den mikrobiellen Gemeinschaften des landwirtschaftlichen Bodens. An Hunderten neuer Bakteriophagen, die dabei entdeckt wurden, sollte untersucht werden, ob Bakteriophagen AMR-Gene tragen, und auf welche Weise sie diese auf die gesamte Gemeinschaft verteilen. Den Ergebnissen zufolge können einige Staphylokokkenstämme – wenn auch langsamer – spezifische Resistenzgene auf Bakterienarten übertragen, die klinisch relevant sind. Dies zeigt die Bedeutung der Vernetzung zwischen Bakteriophagen und Bakterien bei Staphylokokken auf. Die Übertragung erfolgt über Plasmide, d. h. kleine, ringförmige DNA-Abschnitte, die sich außerhalb des bakteriellen Chromosoms befinden. Nach der Sequenzierung und umfassenden bioinformatischen Analyse wurden erstmals in so hoher Zahl neue Bakteriophagen und AMR-Gene enthüllt. Die meisten dieser Bakteriophagen zeigten ein breites Wirtsspektrum, konnten auch klinisch relevante multiresistente Stämme infizieren und trugen verschiedentlich klinisch relevante AMR-Gene. Die Annotation dieser entdeckten AMR-Gene könnte künftig viel dazu beitragen, die genetischen Ursachen von Resistenzbildungen zu klären.
Bedeutung von ROPHARE und Zukunftsaussichten
ROPHARE lieferte völlig neue Erkenntnisse zur mikrobiellen Ökologie und Zusammensetzung in landwirtschaftlich genutzten Böden wie auch zur Rolle von Bakteriophagen für die mikrobielle Ökologie und Evolution. Bezeichnenderweise zeigte die bei Viren als auch Bakterien entdeckte Artenvielfalt, dass die Anwendung landwirtschaftlicher Düngemittel die mikrobielle Zusammensetzung verändern kann. Durch Untersuchung der Dynamik von Ökosystemen, die vom Menschen geschaffen wurden, zeigt ROPHARE, wie durch menschlichen Eingriff der Transfer von AMR-Genen auf tierische und menschliche Mikrobiome gefördert wird. Die Ergebnisse können schließlich auch Ausgangspunkt für neue antimikrobielle Substanzen oder Antibiotikakombinationen sein, die die AMR-Ausbreitung hemmen. Zudem wird mit der Identifizierung und Charakterisierung neuer Bakteriophagen der Weg für die Entwicklung neuer antimikrobieller Strategien und Therapien frei. „Über molekulare Analysen der identifizierten Bakteriophagen konnten wir die vielversprechendsten Kandidaten für antimikrobielle Strategien mit geringem Risiko für die Mobilisierung von AMR-Genen bestimmen“, betont Gómez-Sanz. Derzeit liegt die weltweite Sterblichkeit durch antimikrobielle Resistenzen bei etwa 700 000 Menschen, diese Zahl könnte bis zum Jahr 2050 exponentiell zunehmen. Die Ergebnisse von ROPHARE sollen Interessengruppen bei den erforderlichen Strategien zur Minimierung von Risiken unterstützen. Indem potenzielle Reservoirs von AMR-Genen in der Umwelt enthüllt werden, könnten auch Entscheidungen zur Düngung von Bodensystemen mit Gülle, zum Abwassermanagement und zu anderen Strategien zur Vermeidung der AMR-Ausbreitung erleichtert werden.