Wyśledzono genetyczny komponent chorób autoimmunologicznych
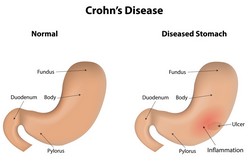
Całogenomowe badania asocjacyjne umożliwiły identyfikację genetycznych czynników chorób autoimmunologicznych, takich jak RZS oraz ChL-C. Mimo to dalsze badania genomiczne były jak do tej pory ograniczone, jako że te allele ryzyka są powiązane z niekodującymi obszarami genomu. Uczestnicy projektu CHIP SEQ RA CD (Identification of regulator variants in rheumatoid arthritis and Crohn’s disease) wykorzystali w badaniach epigenetyczne znaczniki chromatyny. Naukowcy wyodrębnili najistotniejsze znaczniki chromatyny z setek możliwych jej modyfikacji histonowych, które mogą dostarczać zbędnych informacji. Nie tylko scharakteryzowano najodpowiedniejsze znaczniki chromatyny, lecz zidentyfikowano również kluczowe typy komórek, w których można badać geny ryzyka. Identyfikacja najbogatszych w informacje znaczników chromatyny umożliwiła śledzenie odnośnej swoistości komórkowej. Następnym etapem była identyfikacja na poziomie tkankowym, a następnie genetycznym elementów regulacyjnych, na które wpływ miały warianty ryzyka. Reasumując, zespół wyjaśnił rolę wariantów ryzyka w wielu skomplikowanych chorobach, które stanowią obecnie najważniejsze wyzwania medyczne w Europie. To nowatorskie podejście nakierowane jest na RZS, ChL-C, chorobę Parkinsona i cukrzycę typu 2. Wyniki szeroko rozpowszechniono poprzez recenzowane czasopisma i konferencje, oraz udostępniono kod źródłowy stworzonych schematów. Wyniki naukowe projektu dostarczą solidnych podstaw do identyfikacji nowych kaskad molekularnych leżących u podłoża tych złożonych chorób. Może to pomóc odkryć nowe potencjalne geny choroby i umożliwić tworzenie leków celowanych.